mmRNA/CoS-miRy-wstępna analiza oddziaływania z genomem człowieka
Przesłanka ogólna
mmRNA preparatów składa się oprócz kodu białka Kolca (Spike, S) także z sekwencji RNA obecnych w transkryptach innych genów człowieka, które potencjalnie mogą oddziaływać z DNA stając się częścią niekodującego RNA (ncRNA).
I tak:
5′-UTR (ang. 5-prim UnTranslated Region) mRNA szczepionki Pfizer/BioNTech zawiera 5′-UTR ludzkiego genu α-globiny (Rysunek 2A – artykuł źródłowy Xia et al. 2021) ponieważ mRNA α- globiny ulega bardzo szybkiej i wydajnej translacji [jest to dodatkowy czynnik podnoszący wydajność produkcji białka Kolca]. Z kolei koniec 3′-UTR mRNA, zawiera elementy regulujące stabilność mRNA i także pochodzące z ludzkich genów α-globiny i β-globiny. Te elementy często tworzą kompleksy RNA-białko w celu stabilizacji mRNA. 5′-UTR i 3′-UTR genów globiny, po ligacji z innymi mRNA, mogą nadawać stabilność tym innym mRNA. mmRNA-1273 firmy Moderna posiada 110 nt [nt = nukleotydy] 3′-UTR pochodzący z ludzkiego genu α-globiny (HBA1) pomiędzy ostatnim kodonem stop, a początkiem tzw. ogona poli(A). Z kolei 3′-UTR mmRNA firmy Pfizer/BioNTech zawiera segmenty RNA, które działają lepiej niż 3′-UTR mRNA ludzkiej β-globiny. Jeden z nich pochodzi z ludzkiego mitochondrialnego 12S rRNA (mtRNR1), a drugi segment pochodzi z ludzkiego genu AES/TLE5.
Zadaliśmy więc pytanie:
Czy mmRNA preparatów zastosowanych jako rodzaj szczepionki przeciwko Covid-19 może w jakikolwiek sposób oddziaływać z DNA genomowym człowieka po dostarczeniu do komórki?
Z poprzedniej publikacji pt. triDNA wiemy, że takie oddziaływania są możliwe i zachodzą w komórce pomiędzy ogólnie definiowanym ncRNA czyli non-coding RNA, którego szczególną frakcją jest tzw. lncRNA czyli long non-coding RNA. Jak wiadomo mmRNA preparatów jest cząsteczką kodującą informację o zmodyfikowanym białku Kolca wirusa SARS2, a zatem na razie nie bierzemy pod uwagę możliwości bezpośredniego oddziaływania tego mmRNA z DNA genomu w funkcji lncRNA. Jednak ncRNA obejmuje także inne klasy RNA w tym m. in. microRNA (miRNA, miR), a ten rodzaj RNA powstaje z mRNA w komórce.
Zapytaliśmy więc czy istnieją już analizy wykazujące, że mmRNA preparatów może być źródłem microRNA i jeśli tak to jakiego rodzaju jest to / są to miR’y?
W pracy Fujii 2021 czytamy, że szczepionka mRNA przeciwko Covid-19 z sekwencjami białka Kolca (S) może wytwarzać microRNA (miRNA). Nie ma jednak badań takiego miRNA pod kątem bezpieczeństwa szczepionek przeciwko Covid-19. Dlatego ocena miRNA ze szczepionek jest konieczna do zarządzania ryzykiem szczepień. I tak, pre-miRNA przewidywano w badaniu Fujii 2021 na podstawie sek wencji RNA białka S wirusa SARS-CoV-2 . Znaleziono szereg cząsteczek microRNA, które w większości posiadają odpowiednie homologii wśród znanych już wcześniej ludzkich miR’ów.
W badaniu uwzględniono także fakt, że Ψ [pseudourydyna] może tworzyć wiązania z adeniną, a aktywność Ψ w zależnym od RNA wyciszaniu genów jest nieregularnie zmieniana zarówno przez położenie Ψ w sekwencji RNA, jak i ogólną z a wartość Ψ w RNA. Preparaty mmRNA zostały „nasycone” Ψ w znacznym stopniu i dlatego jest też możliwe, że miRNA pochodzące z BNT162b2 miałoby dużą siłę oddziaływania właśnie ze względu na zawartość N1-metylo-Ψ [wersji pseudourydyny obecnej w preparatach mmRNA] w cząsteczkach microRNA powstającego z preparatów mmRNA. Ostatecznie w pracy Fujii 2021 przeprowadzono więc analizę w celu przewidzenia funkcji miRNA pochodzących bezpośrednio z BNT162b2.
W wyniku tej analizy, wyodrębniono następujące miR’y i ich homologi u ludzi:
- CovS-miR-3 / homolog miRNA i stopień homologii: hsa-miR-4504 / 75%
- CovSmiR-14 / hsa-miR-5590-5p / 75%
- CovS-miR-21 / hsa-miR-142-3p / 88%
- CovS-miR-48.1 / hsa-miR-4512 / 75%
- CovS-miR-50.3 / hsa-miR-4793-3p / 100%
- CovS-miR-55 / hsa-miR-1265 / 88%
- CovS-miR-62 / hsa-miR-6770-5p / 50%
Te miR’y pochodzą z mmRNA preparatów i już w pracy Fujii 2021 zostały dla nich wyodrębnione określone geny, z którymi te microRNA oddziałują na drodze epigenetycznej regulacji ekspresji genów, co oznacza wyciszanie aktywności tych genów poprzez regulowanie procesu degradacji mRNA transkrybowanego z tych genów. Oznacza to, że tak zidentyfikowane miR’y wywodzące się z mmRNA preparatów już są dowodem na pośrednią funkcję mmRNA preparatów w modulowaniu aktywności genetycznej komórki.
My jednak zadaliśmy jeszcze inne pytanie:
Wiemy już, że te wyselekcjonowane miR’y mogą oddziaływać w klasycznym schemacie epigenetycznej, post-transkrypcyjnej regulacji ekspresji genów typu microRNA-mRNA-miRISC, ale czy microRNA pochodzące z preparatów może także bezpośrednio oddziaływać z DNA poprzez mechanizm TFO czyli Triplex Forming Oligonucleotides, w którym krótki fragment RNA (lub pojedynczoniciotwego DNA, ssDNA) wiąże się z DNA parami Hoogsteena, a nie Watsona-Cricka wewnątrz tzw. dużego rowka DNA?
Nie od dzisiaj wiadomo, że helisa podwójnoniciowego DNA (dsDNA) jest zdolna do tworzenia struktur tzw. potrójnej helisy i że microRNA o odpowiedniej długości i sekwencji tworzą struktury takiej potrójnej helisy z sekwencjami dsDNA, tzw. triplexów/tripleksów. Geny zawierające sekwencje sprzyjające tworzeniu tripleksów przy udziale microRNA są dodatnio skorelowane z ekspresją cząsteczek microRNA ukierunkowanych na sekwencje generujące tripleks [patrz literatura].
A zatem w jaki sposób sprawdzić czy wyselekcjonowane wcześniej miR’y w pracy Fujii 2021 oddziałują z genomowym DNA poprzez potencjalne formowanie triplexów DNA i jeśli tak to w jakich miejscach takiego genomowego DNA kompleksy typu TFO DNA (triDNA) powstają?
Istnieje baza danych i narzędzie bio-informatyczne, które na taką analizę pozwala [niestety i jak zwykle… nie w Polsce]. Narzędzie i baza danych o nazwie RNA-Chrom analizuje i klasyfikuje oddziaływania RNA z DNA chromosomów w ramach tzw. interactomu RNA-chromatyna.
W pracy Ryabykh et al. 2023 czytamy, że:
RNA odgrywają ważną rolę w procesach biologicznych wpływających na różne poziomy organizacji układów żywych: od komórkowego (regulacja ekspresji genów, przebudowa i utrzymanie struktury chromatyny, ko-transkrypcyjna supresja transpozonów, splicing, po-transkrypcyjne modyfikacje RNA, itp.) do poziomu populacji komórek, a nawet organizmów (rozwój, starzenie się, nowotwory, choroby układu krążenia i wiele innych). Rozwój i tworzenie wzajemnie uzupełniających się baz danych, które będą agregować, ujednolicać i strukturyzować
różne typy danych, może pomóc w osiągnięciu poziomu systemowego badania niekodujących RNA [ncRNA]. Baza danych RNA-Chrom, zawiera współrzędne miliardów kontaktów dla ludzkich i mysich RNA z chromatyną.
Interfejs internetowy (https://rnachrom2.bioinf.fbb.msu.ru) proponuje dwa podejścia do analizy interakcji RNA–chromatyna.
Pierwsze podejście pozwala na sprawdzenie czy interesujący RNA styka się z chromatyną, a jeśli tak, to z jakimi genami lub loci DNA [większy obszar DNA niż jeden czy nawet kilka genów]?
Drugie podejście pozwala dowiedzieć się, które RNA mają kontakt z danym obszarem DNA interesującym użytkownika (i w związku z tym najprawdopodobniej uczestniczą w regulacji aktywności tego DNA) i jeśli takowe istnieją, jaki jest charakter ich interakcji?
Do wstępnej analizy wybraliśmy zidentyfikowane wcześnie miR’y, o których wiemy, że powstają z mmRNA preparatów. Są nimi:
CovS-miR-50.3 wykazujący 100% homologii z ludzkim microRNA hsa-miR-4793-3p
CovS-miR-21 wykazujący 88% homologii z ludzkim microRNA hsa-miR-142-3p
CovS-miR-55 wykazujący 88% homologii z ludzkim microRNA hsa-miR-1265
Badaliśmy ludzkie homologii tych miR’ów wyodrębnionych z mmRNA preparatów ponieważ baza danych nie zawiera dokładnych sekwencji tych CovS-miR’ów.
Wstępne wyniki:
Poddając analizie ludzki microRNA hsa-miR-4793-3p CovS-miR-5 0.3 wykazujący 100% homologii z CovS-miR-50.3, nie wykryliśmy żadnych punktów kontaktu z chromosomowym DNA. Zatem najprawdopodobniej CovS-miR-50.3 microRNA powstający z mmRNA preparatów nie oddziałuje z chromosomalnym DNA ale jest najprawdopodobniej zaangażowany w post- transkrypcyjną regulację aktywności genetycznej komórki w mechanizmie microRNA-mRNA-miRISC.
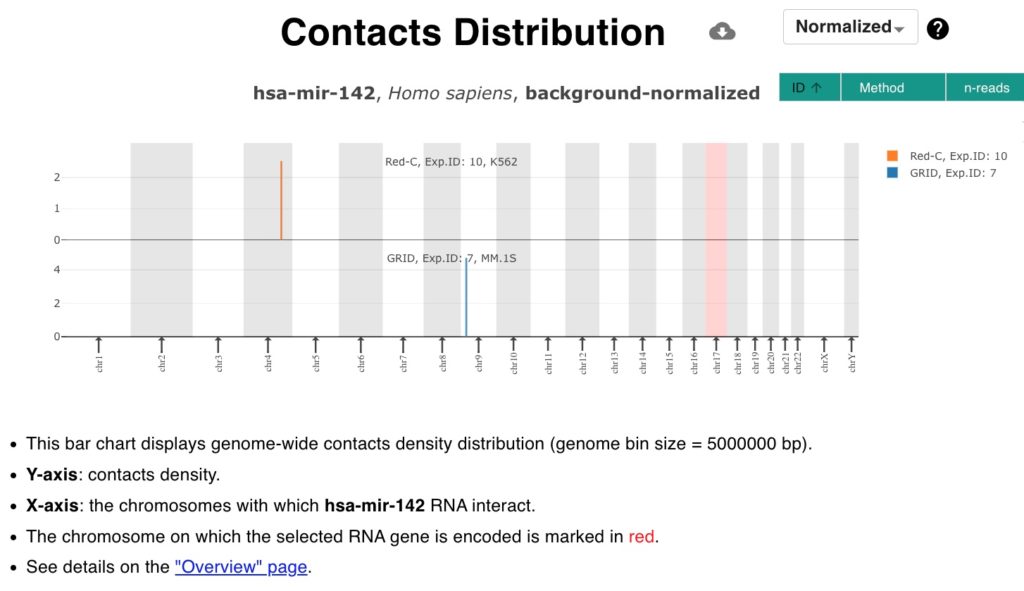
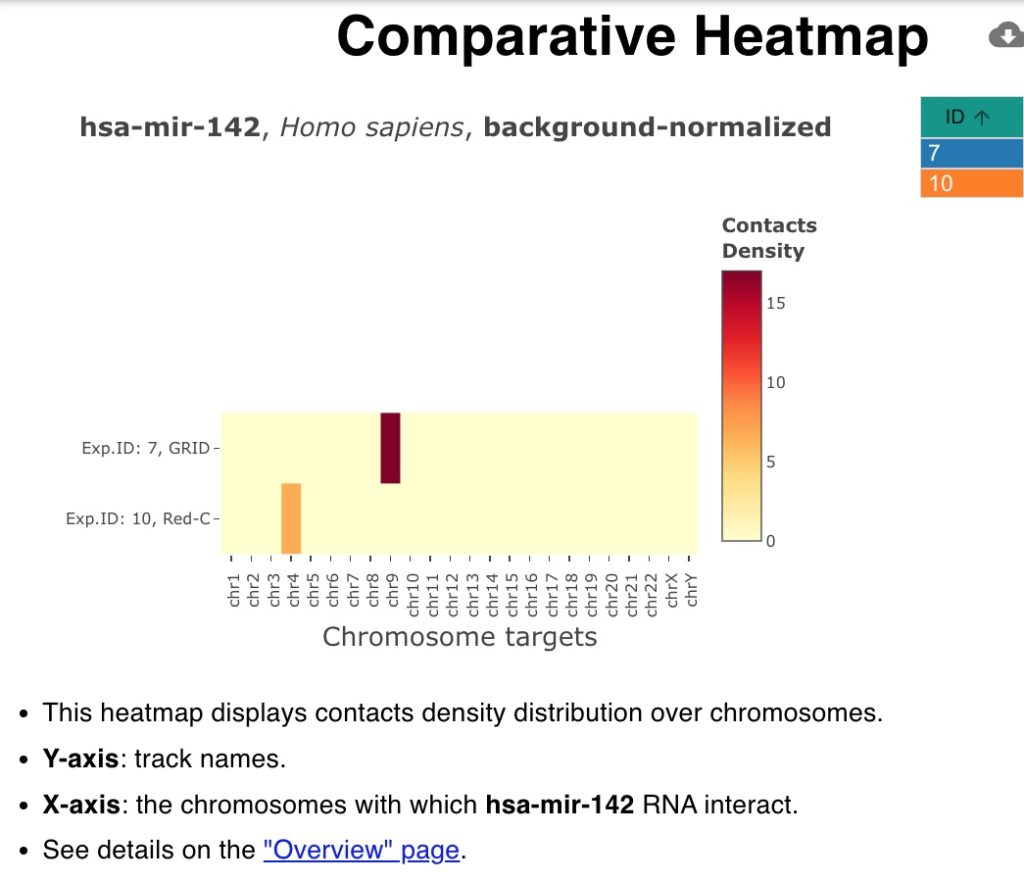
Dalej analizując microRNA hsa-miR-142-3p, wykazujący 88% homologii z CovS- miR-21, otrzymaliśmy następujący wynik:
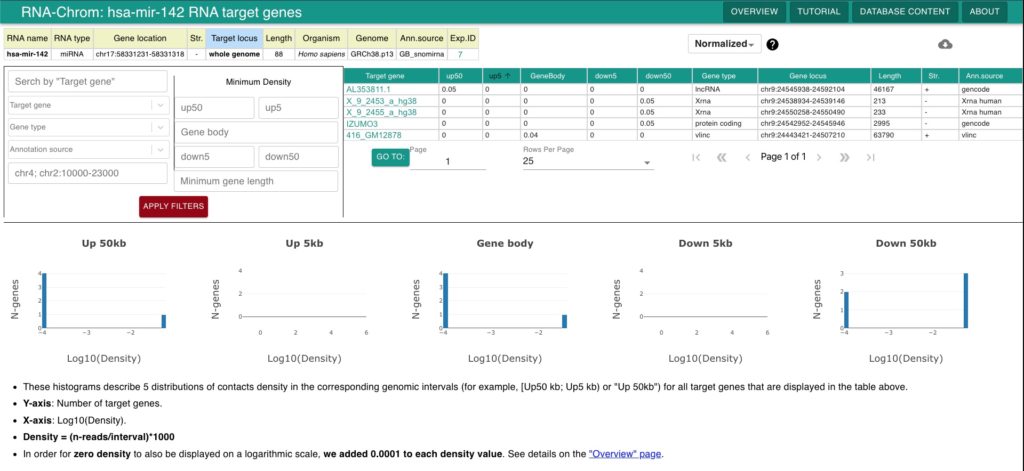
hsa-miR-142 (3p) oddziałuje z genami na chromosomie 4 i 9, przy czym oddziaływanie na chromosomie 9 jest silniejsze i obejmuje:
- Jeden gen dla lncRNA AL35311.1 regulującego aktywność nawet kilkuset innych genów w komórce.
- Dwa geny dla Xrna [5 ’–3 ’ exoribonuclease] X_9_2453_a_hg38 oraz
X_9_2455_a_hg38, których zablokowanie może mieć stabilizujący efekt na większości mRNA zależnych od tych Xrna. - Jeden gen kodujący białko IZUMO3 z dysfunkcją, którego związana jest choroba CDL kości czaszki z łamliwością kości, która charakteryzuje się niską gęstością mineralną kości, licznymi złamaniami kręgosłupa i kości rozpoczynającymi się w dzieciństwie oraz sklerotycznymi zmianami w kościach czaszki. U niektórych poważniej dotkniętych osób stwierdza się złamania w okresie noworodkowym, znaczny niski wzrost, wyraźne stwardnienie czaszki i dysplazję kręgosłupa (CDLSMD).
- Jednym genem vlincRNA 416_GM12878 czyli very long intergenic non-coding
RNA. vlincRNA regulują wiele genów kodujących białka zaangażowane głównie w funkcje związane z RNA i rozwojem, cykl komórkowy i adhezję komórkową poprzez mechanizm obejmujący bliskość vlincRNA i ich celów w jądrze [genów]. Typowe vlincRNA mogą być zarówno pozytywnym, jak i negatywnym regulatorem i regulować wiele genów zarówno w układzie trans-jak i cis- [na tym samym chromosomie]. vlincRNA i ich sieci regulacyjne potencjalnie reprezentują nowe składniki odpowiedzi na uszkodzenie DNA [patrz literatura]
Dalej analizując kolejny homolog microRNA hsa-miR-1265 dla CovS-miR-55 wykazujący 88% zgodności stwierdziliśmy, że również i ten miR nie wykazuje kontaktu z DNA genomowym w analizie opartej o narzędzie i bazę danych interactomu RNA-chromatyna. Postanowiliśmy więc sprawdzić pozostałe miRy wyselekcjonowane wcześniej jako microRNA postające z mmRNA preparatów przeciwko Covid-19. Nie stwierdziliśmy aby jakikolwiek pozostały miR oddziaływał z DNA chromosomów.
W tej sytuacji przeanalizowaliśmy również nieco słabsze oddziaływanie hsa- miR-142(3p) na chromosomie 4. I tak na tym chromosomie homolog CovS-miR-21 oddziałuje z 7 genami, z których 2 są genami dla lncRNA, 3 dla Xrna i 2 dla snRNA.
Wnioski:
Bez zagłębiania się w dalszą analizę możemy powiedzieć, że szczególnie jeden rodzaj microRNA powstający z mmRNA preparatów przeciwko Covid-19, a mianowicie CovS-miR-21, którego ludzkim homologiem kodowanym na chromosomie 17 jest hsa-miR-142(3p) może oddziaływać z genomowym DNA w jądrze komórki najprawdopodobniej poprzez formowanie struktury tripletu DNA. Oddziaływanie to ma prawdopodobnie charakter blokujący funkcje genów, z którymi to microRNA oddziałuje szczególnie jeżeli miejsce kontaktu RNA:DNA znajduje się wewnątrz genu [gene body], a więc w sekwencji samego kodu, a nie w obrębie regionów regulatorowych gdzie potencjalnie takie oddziaływanie mogłoby mieć charakter wspomagający ekspresję danego genu.
Z uwagi na to, że większość oddziaływań miR-142(3p) zarówno na chromosomie 4 jak i 9 znajduje się w obrębie genów związanych z regulacyjną rolą RNA w ekspresji genów i oddziaływanie to jest najprawdopodobniej hamuj ą ce i/lub wytłumiaj ą ce to należy założyć, że w przypadku oddziaływania z genami kodującymi lncRNA – negatywny wpływ preparatów mmRNA zostanie przeniesiony na zaburzenia ekspresji co najmniej kilkuset genów regulowanych przez to lncRNA, wytłumione miR-142(3p). Nie analizowaliśmy już tutaj jakie to są geny. Niemniej jednak ich liczba na obu chromosomach może sięgać nawet kilkuset różnych genów, co z kolei podkreśla jak ważną rolę spełnia lncRNA w regulacji ekspresji genów.
Innymi genami na jakie wpływa układ CovS-miR-21 / miR-142(3p) są geny exorybonukleazy (Xrna) co należy interpretować jako najprawdopodobniej podniesienie żywotności wszystkich cząsteczek mRNA w komórce, zależnych od tych konkretnych exorybonukleaz. Potencjalnie jest to efekt pozytywny gdyż podniesienie okresu półtrwania mRNA opóźnia proces molekularnego starzenia się komórki, ale z drugiej strony zbyt długi czas trwania mRNA komórkowego może mieć także negatywny skutek w postaci redukcji ekspresji genów i/lub blokowania systemu translacji-fałdowania-eksportu białek komórki w przypadku kiedy zbyt często poszczególne cząsteczki niezdegradowanego mRNA wchodzą w cykle translacji na rybosomach.
Istotne są też oddziaływania CovS-miR-21 / miR-142 (3p) z snRNA [small nuclear RNA]. Ten typ RNA jest kluczowym składnikami tzw. spliceosomu, który katalizuje składanie pre-mRNA. snRNA oddziałują z wieloma białkami, tworząc w jądrze komórkowym kompleksy RNA-białko, zwane małymi jądrowymi rybonukleoproteinami (snRNP). Jeżeli oddziaływanie CovS-miR-21 / miR-142 (3p) zgenami dla co najmniej dwóch snRNA ma charakter blokujący to upośledzone zostanie prawidłowe składanie komórkowego mRNA co może prowadzić do patologii związanych z tzw. alternatywnym splicningiem i powstawaniem izoform danego białka/ek. Przykładem takich patologii mogą być nowotwory, choroby układu krążenia i różne neuropatie, takie jak choroby Alzheimera, Parkinsona i Huntingtona, a także schizofrenia i wrodzony zespół miasteniczny. Być może nie bez znaczenie jest tutaj także fakt, że zakodowany w DNA mRNA kodujący białko Kolca można złożyć w taki sposób, że transbłonowa domena białka Kolca
zostanie utracona, dzięki czemu białko to będzie wydzielane z komórek niemal w całości co może leżeć u podstaw mechanizmu, który mógłby wyjaśnić zdarzenia zakrzepowo-zatorowe występujące po szczepionkach przeciwko COVID opartych na DNA [patrz literatura].
I wreszcie zidentyfikowane oddziaływanie CovS-miR-21 / miR-142 (3p) z jednym genem kodującym białko IZUMO3, którego dysfunkcja u ludzi wiąże się z uszkodzeniem kośćca jest jeszcze o tyle zastanawiające, że np. u myszy białka z tej rodziny są zasadniczo białkami fuzyjnymi plemnika. Podczas zapłodnienia wiążą się one ze swoim receptorem na komórce jajowej (Juno), w celu ułatwienia rozpoznania i połączenia obu gamet. Badania wykazały, że samce myszy pozbawionych genu Izumo są bezpłodne, ponieważ ich plemniki nie są w stanie połączyć się z błoną oocytu . Myszy Izumo -/- [ -/- / tak oznacza się tzw. knock-out czyli „wybicie” oby apelli danego genu z chromosomu] wytwarzały plemniki o prawidłowej morfologii, które były w stanie przeniknąć przez osłonkę przezroczystą, ale nie mogły połączyć się z jajami. W eksperymentach in vitro u ludzi, białka z rodziny IZUMO również okazały się niezbędne do fuzji ludzkich gamet. Nie bez znaczenia jest także fakt, że szczepienie białkiem Izumo lub jego cDNA (!!) powoduje znaczne zmniejszenie płodności samic myszy. Przeciwciała przeciwko ludzkiemu białku IZUMO hamują penetrację ludzkich plemników do oocytu [patrz literatura].
Istnieje zatem możliwość, że CovS-miR-21 /miR-142(3p) oddziałujący z genem IZUMO3 może również oddziaływać z innymi wysoce homologicznymi genami z rodzimy białek IZUMO i jeśli tak to nie jest wykluczone, że miałoby to bardzo negatywny wpływ na rozrodczość człowieka mimo, iż morfologicznie wszystkie parametry spermy byłyby prawidłowe . Brak białka IZUMO w komórkach spermy na skutek zablokowania jego ekspresji poprzez uformowanie potencjalnej struktury triDNA w obrębie genu/ów z rodziny IZUMO przez microRNA powstające
z mmRNA preparatów przeciwko COVID, byłoby raczej trwałe lub długotrwałe i stałoby się przyczyną drastycznego spadku liczby urodzeń obok efektów związanych z uszkodzeniem kości czaszki i kośćca w przypadkach niektórych urodzeń. Lekarze położnicy mogą te hipotezy zweryfikować zarówno badaniem stanu rozwoju kośćca u dzieci urodzonych z zaszczepionych rodziców jak i obserwacją procesu zapłodnienia in vitro spermą pochodzącą od zaszczepionych mężczyzn.
Podsumowując, zaprezentowane tutaj analizy i wnioski są wstępne i bardzo ogólne. Ich rolą jest nakreślenie szerszej perspektywy spojrzenia na preparaty mmRNA i ich rolę w całej fizjologii komórki, a nie tylko w jednym jej aspekcie związanym z translacją mRNA prowadzącą do powstania białka Kolca w celu wygenerowania odpowiedzi immunologicznej. mRNA jest biologicznie najbardziej wszechstronną i reaktywną cząsteczką występującą w komórkach i wielkim oraz potencjalnie fatalnym w skali całej populacji błędem jest przypuszczenie, że mmRNA preparatów ma tylko i wyłącznie jedną rolą w komórce.
Nie ma bo po prostu taka jest natura RNA i przedstawiona tutaj wstępna i ogólna analiza wskazuje tylko niektóre kierunki, w których powinny się rozwijać dalsze badania preparatów mmRNA, jeżeli chcemy aby w przyszłości preparaty te rzeczywiście leczyły, a nie… okaleczały, a nawet prowadziły do wymarcia gatunku co de facto może mieć miejsce jeżeli np. hipotezy o negatywnym wpływie mmRNA preparatów na rozród człowieka za pośrednictwem np. struktur triDNA w obrębie rodziny genów IZUMO – okazałyby się prawdziwe i długoterminowe.
Nie można także zapominać, że preparaty mmRNA zawierają rówież zanieczyszczenia DNA zarówno w formie nienaruszonego plazmidowego DNA (plDNA) jak i fragmentów podwójnoniciowego DNA (dsDNA). Z ostatnich odkryć może jednak wynikać, że oile samo, nienaruszone plDNA może niestanowić bardzo poważnego zagrożenia, o czym piszemy w naszym innym artykule na patreon.com/pradma, to fragmenty plDNA mogą już być dodatkowym problemem związanym z integracją tych fragment ów z genomowym DNA jak również wykorzystaniem ich w procesach tworzenia triplexów DNA.
Należy też pamiętać, że przeprowadzona tutaj ogólna analiza odbywała się przy użyciu ludzkich homologów, zidentyfikowanych microRNA powstających z mmRNA preparatów. Tylko w jednym przypadku homologia ta wynosiła 100% i nie wykazała żadnego oddziaływania pomiędzy tym microRNA, a DNA chromosomów. Zakres homologii w pozostałych przypadkach wahał się od 50% do 88%, ale tylko w jednym przypadku o homologii 88% odkryto potencjalne oddziaływania z DNA chromosomów.
Nie wolno jednak ignorować faktu, że wyselekcjonowane microRNA zawierają w swoich sekwencjach pseudourydynę w formie N1-metylowanej, która oddziałuje silniej niż uracyl z DNA. Tym samym nie jest wykluczone, że badając bezpośrednio sekwencję microRNA, którego homolog wykazał oddziaływanie z DNA nawet tylko przy 88% homologii, mogłoby się okazać, że obecność pseudourydyny powoduje jeszcze silniejszą i bardziej specyficzną interakcję tego szczególnego microRNA z DNA chromosomów.
Zaprezentowana tutaj analiza nie stanowi żadnej formalnej podstawy do szczegółowego wnioskowania o dalszych, nietypowych i potencjalnie szkodliwych skutkach obecności mmRNA preparatów w komórkach osób, które przyjęły tzw. szczepionki przeciwko COVID-19. Uważamy jednak, że wskazuje ona kierunek w jakim powinny się potoczyć szeroko zakrojone badania preparatów mmRNA.
Niestety jesteśmy świadomi, że wszelkie instytucje państwa, a więc uniwersytety mimo iż posiadają dość daleko idącą niezależność od struktur rządowych, ale i państwowe ośrodki badawcze – nie podejmą takich badań będąc wcześniej aktywnie zaangażowanymi w marketing preparatów mmRNA i przekonywanie o ich absolutnym bezpieczeństwie. Z tego powodu przedstawione tutaj analizy pozostaną tylko spekulacjami aż do momentu, w którym ktoś w krajach bardziej obywatelskich, demokratycznych i przed wszystkim wolnych niż obecna Polska, dysponując odpowiednimi środkami i zespołem przeprowadzi podobne ale już o wiele bardziej szczegółowe analizy weryfikując zaprezentowany przez nas kierunek badań preparatów mmRNA.
Według naszej wiedzy jest to także pierwsza, przynajmniej w Polsce, próba analizy preparatów mmRNA pod kątem ich oddziaływania z genomowym DNA.
Literatura:
Xia X. Detailed Dissection and Critical Evaluation of the Pfizer/BioNTech and Moderna mRNA Vaccines. Vaccines (Basel). 2021 Jul 3;9(7):734. doi: 10.3390/vaccines9070734.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8310186/
Fujii YR. Quantum microRNA Assessment of COVID-19 RNA Vaccine: Hidden Potency of BNT162b2 SASR-CoV-2 Spike RNA as MicroRNA Vaccine. March 05, 2021; Published: March 16,
https://crimsonpublishers.com/aics/fulltext/AICS.000552.php
Paugh SW, Coss DR, Bao J, Laudermilk LT, Grace CR, Ferreira AM, Waddell MB, Ridout G, Naeve D, Leuze M, LoCascio PF, Panetta JC, Wilkinson MR, Pui CH, Naeve CW, Uberbacher EC, Bonten EJ, Evans WE. MicroRNAs Form Triplexes with Double Stranded DNA at Sequence-Specific Binding Sites; a Eukaryotic Mechanism via which microRNAs Could Directly Alter Gene Expression. PLoS Comput Biol. 2016 Feb 4;12(2):e1004744.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4742280/
Ryabykh GK, Kuznetsov SV, Korostelev YD, Sigorskikh AI, Zharikova AA, Mironov AA. RNA-Chrom: a manually curated analytical database of RNA-chromatin interactome. Database (Oxford). 2023 Apr 24;2023:baad025. doi: 10.1093/database/baad025. Erratum in: Database (Oxford). 2023 Jul 1;2023.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC10205464/
Cao, H., Xu, D., Cai, Y. et al. Very long intergenic non-coding (vlinc) RNAs directly regulate multiple genes in cis and trans. BMC Biol 19, 108 (2021).
https://doi.org/10.1186/s12915-021-01044-x
Kowarz E, Krutzke L, Külp M, Streb P, Larghero P, Reis J, Bracharz S, Engler T, Kochanek S, Marschalek R. Vaccine-induced COVID-19 mimicry syndrome. Elife. 2022 Jan 27;11:e74974. doi: 10.7554/eLife.74974. https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8846585/
Inoue, N., Ikawa, M., Isotani, A. et al. The immunoglobulin superfamily protein Izumo is required for sperm to fuse with eggs. Nature 434, 234–238 (2005). https://doi.org/10.1038/nature03362
Rajesh K. Naz. Vaccine for human contraception targeting sperm Izumo protein and YLP12 dodecamer peptide . Protein Science. Volume 23, Issue 7. July 2014. Pages 857-868.
https://onlinelibrary.wiley.com/doi/full/10.1002/pro.2476

Zobacz także
Wywiad ze sztuczną inteligencją
Straceni dla nieba…
Berezwecz – kolejny Katyń