Od dawna sugerujemy, że preparaty mmRNA mogą albo bezpośrednio uczestniczyć w regulacji ekspresji genów lub pośrednio poprzez mechanizm zależny od tzw. lncRNA (ang. long non-coding RNA).
Ostatnio wyłoniły się pewne wnioski z napływających badań w powiązanych obszarach, które wspierają takie wciąż spekulacje na tym etapie, gdyż nie mając dostępu do dedykowanych projektów badawczych w tym kierunku w ramach niezależnego instytutu badawczego, a żadna instytucja państwa tego nie zweryfikuje ponieważ potwierdzenie takiej funkcji mmRNA będzie dramatem dla ludzkości – nie możemy tego zweryfikować ani bio-informatycznie, ani tym bardziej eksperymentalnie. Możemy tylko zaprezentować nasze hipotezy i czekać aż ktoś, gdzieś, kiedyś je zweryfikuje w krajach bardziej wolnych niż obecna Polska.
Poniżej prezentujemy tylko wstęp do bardziej rozległej hipotezy dotyczącej roli mmRNA w modulowaniu aktywności genetycznej komórki. W dalszych publikacjach będziemy prezentowali kolejne elementy tej hipotezy. Od razu zaznaczamy także, że wszystko o czym będziemy pisać w ramach tej hipotezy jest absolutnym horyzontem zdarzeń biologii molekularnej mimo iż poszczególne elementy znane są od wielu lat to jednak szczegółowe mechanizmy są poznawane dopiero teraz. Wynika to głównie z tego, że cząsteczki RNA należą do najstarszych ewolucyjnie cząsteczek aktywnych biologicznie i w związku z tym dzisiaj wszystkie ich funkcje – a są to naprawdę funkcje biologicznie podstawowe – mają charakter bardzo subtelny tzn. nie są oczywiste wprost i można je zidentyfikować dopiero po zastosowaniu bardzo wyrafinowanych analiz i technik biologii molekularnej co wymaga czasu, rozwoju nowych metod analitycznych i olbrzymiej wyobraźni ponieważ na tak głębokim poziomie molekularnym, podobnie jak w fizyce kwantowej (świat RNA jest o krok od zjawisk kwantowych) wszystkie mechanizmy nie są od razu oczywiste.
A zatem rysunek jaki dołączyliśmy do niniejszego artykułu przedstawia tzw. TRIPLEX DNA czyli strukturę normalnego fizjologicznie B-DNA (podwójna helisa), do której w tzw. wielkim/dużym rowku DNA przyłączona jest dodatkowa nić. W warunkach fizjologicznych komórki będzie to najczęściej nic ssRNA (ang. single-stranded RNA), ale może to być nic ssDNA lub nawet… tzw. PNA czyli Peptide-Nucleic Acid.
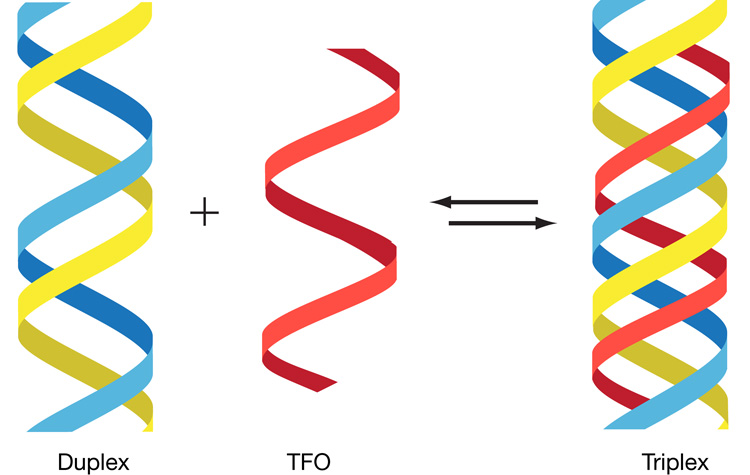
Na rysunku w docelowym dupleksie nici purynowa i pirymidynowa są pokazane odpowiednio w kolorze niebieskim i żółtym. TFO (ang. Triplex-Forming Oligonucleotide RNA lub rzadziej DN), które wiąże się z nicią bogatą w purynę docelowego dupleksu przez duży rowek duplexu DNA, zaznaczono na czerwono. (szczegóły – patrz: literatura dodatkowa, Jain et al. 2008). Nie będziemy teraz rozpisywać się nad szczegółami tego rysunku – napiszemy o tym później omawiając mechanizm tworzenia się takich triplexów DNA (triDNA). Tutaj chodzi tylko o to, aby zobrazować jak wygląda takie triDNA gdzie czerwona nicią może być nie tylko oligonukleotyd (krótka sekwencja RNA/DNA), ale także fizjologicznie funkcjonalne lncRNA.
W jednej z prac czytamy:
Zidentyfikowaliśmy 727 lncRNA o różnej ekspresji (153 o zwiększonej) u pacjentów z VBT (zakażenia przełomowe po szczepieniu). Obejmują one modulację odp. immunolo. i odp. zapalnej w VBT. Analiza koekspresji lncRNA-mRNA ujawniła, że 34 lncRNA oddziałują z 267 mRNA. Zaobserwowaliśmy również większą liczebność Alu, LINE1 i LTR w grupie aktywnie oddziałujących lncRNA u pacjentów z VBT. Te lncRNA mają wyższą interakcję z regionem promotora genów z tego samego TAD (domena powiązana topologicznie) w porównaniu z lncRNA nieoddziałującymi. Dodatkowo promotor genów, z którymi oddziałują lncRNA jest wzbogacony w elementu Alu i LINE1.
i dalej…
Homologiczna interakcja między lncRNA i genami posiadającymi domeny TAD jest kontrolowana przez elementy Alu i LINE1. Jest to możliwy mechanizm tłumienia wrodzonych odpowiedzi immunologicznych/zapalnych oraz tłumienia aktywacji adaptacyjnej odpowiedzi immunologicznej za pośrednictwem lncRNA. Może to wyjaśniać przełomowe infekcje SARS-CoV-2 u osób VBT.
Dane te pokazują także trójwymiarową regulację genów za pośrednictwem lncRNA oraz powtarzalnych elementów genomu (Alu, LINE1) jako kolejny możliwy sposób modulowania regulacji immunologicznej po szczepieniu mmRNA.
W innej pracy czytamy…
lncRNA mogą łączyć się z kompleksami modyfikującymi histony, a następnie atakować DNA. Na przykład, łącząc się z kompleksami modyfikującymi histony, forkhead box F1 (FOXF1) przylegający do niekodującego rozwojowego regulatorowego RNA (FENDRR) zakotwicza się do docelowych fragmentów promotora. Antysensowny RNA transkryptu HOX (HOTAIR) zajmuje również docelowe, podwójnie DNA (dsDNA). Dodatkowo lncRNA transkrybowany z mniejszego promotora genu ludzkiej reduktazy dihydrofolianowej (DHFR) łączy się z kluczowym regionem regulatorowym genu gospodarza w modelu potrójnej helisy. Te potrójne struktury mogą służyć jako specyficzny mechanizm rozpoznawania między lncRNA, a genomowym DNA… [np. dla czynników transkrypcyjnych, które bez asysty lncRNA nie mogłyby aktywować ekspresji danego genu (!)]
Innymi słowy lncRNA lub odpowiednio zmodyfikowane RNA (lub DNA lub PNA) o określonej długości nie mniejszej niż 200 nukleotydów – to jest graniczna długość m. in. definiujące lncRNA – może zostać dostarczone do jądra komórki gdzie po uformowaniu triDNA umożliwi dostęp określonych czynników transkrypcyjnych do inaczej niedostępnego genu. Może jednak być także odwrotnie – takie lncRNA (choć bardziej jest to prawdopodobne z krótszymi fragmentami oligo ncRNA – ncRNA = non-coding RNA co nie oznacza, że takie RNA nie może zawierać w sobie fragmentu jakiejś ramki odczytu dla jakiegoś białka. Oznacza to tylko, że generalnie takiemu ncRNA, którego lncRNA jest bardziej specyficznym typem – nie przyznaje się roli nośnika jakiejś informacji o białku) może zablokować ekspresję określonego genu czyniąc promotor takiego genu niedostępnym dla czynników transkrypcji poprzez uformowanie w nim triDNA blokującego dostęp tych czynników do promotora… Tripleksy utworzone przez lncRNA i genomowy DNA mogą zdecydowanie skutkować specyficznością modulowania aktywności genetycznej. (patrz: literatura dodatkowa)
Podsumowując:
– istnieje biologicznie zdefiniowana możliwość na co najmniej pośredni wpływ preparatów mmRNA na modulację aktywności genów za pośrednictwem co najmniej 153 lncRNA, których ekspresja jest zwiekszona po przyjęciu preparatów.
– modulowanie funkcji genomu za pośrednictwem lncRNA indukowanym preparatami mmRNA jest możliwym mechanizmem tłumienia wrodzonych odpowiedzi immunologicznych/zapalnych oraz tłumienia aktywacji adaptacyjnej odpowiedzi immunologicznej co może wyjaśniać pojawianie się licznych wariantów. ucieczki immunologicznej u osób zaszczepionych mmRNA
– lncRNA tworzy struktury triplexu DNA ułatwiająca lub blokujące dostęp czynników transkrypcji do promotorów określonych genów z którymi takie lncRNA odziałuje
– spekulujemy, że również samo mmRNA preparatów i/lub jego fragmenty i/lub fragmenty zanieczyszczającego te preparaty plazmidowego DNA i/lub mRNA syntetyzowanego w jądrze komórki na bazie plazmidowego DNA, które tam wniknęło mogą oddziaływać z DNA formując specyficzne lub niespecyficzne triDNA
Literatura:
Chattopadhyay P, Mishra P, Mehta P, Soni J, Gupta R, Tarai B, Budhiraja S, Pandey R. Transcriptomic study reveals lncRNA-mediated downregulation of innate immune and inflammatory response in the SARS-CoV-2 vaccination breakthrough infections. Front Immunol. 2022 Nov 18;13:1035111. doi: 10.3389/fimmu.2022.1035111.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9716354/
Literatura dodatkowa:
Jain A, Wang G, Vasquez KM. DNA triple helices: biological consequences and therapeutic potential. Biochimie. 2008 Aug;90(8):1117-30. doi: 10.1016/j.biochi.2008.02.011. Epub 2008 Feb 21. Erratum in: Biochimie. 2018 Mar 23;:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2586808/
Li T, Mo X, Fu L, Xiao B, Guo J. Molecular mechanisms of long noncoding RNAs on gastric cancer. Oncotarget. 2016 Feb 23;7(8):8601-12. doi: 10.18632/oncotarget.6926.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4890990/
—-
Grupa Pradma

Zobacz także
Berezwecz – kolejny Katyń
Covid? Na pewno nie o to chodziło…
Zapomniany obóz NKWD w Ciechanowie